¿Cómo puedo trazar con 2 ejes y diferentes?
Me gustaría superponer dos diagramas de dispersión en R para que cada conjunto de puntos tenga su propio (diferente) eje y (es decir, en las posiciones 2 y 4 de la figura) pero los puntos aparecen superpuestos en la misma figura.
Es posible hacer esto con plot?
Editar Código de ejemplo que muestra el problema
# example code for SO question
y1 <- rnorm(10, 100, 20)
y2 <- rnorm(10, 1, 1)
x <- 1:10
# in this plot y2 is plotted on what is clearly an inappropriate scale
plot(y1 ~ x, ylim = c(-1, 150))
points(y2 ~ x, pch = 2)
6 answers
Actualización : Material copiado que estaba en el wiki de R en http://rwiki.sciviews.org/doku.php?id=tips:graphics-base:2yaxes , enlace ahora roto: también disponible desde la wayback machine
Dos ejes y diferentes en la misma parcela
(algo de material originalmente por Daniel Rajdl 2006/03/31 15: 26)
Tenga en cuenta que hay muy pocas situaciones en las que sea apropiado usar dos escalas diferentes en la misma parcela. Es muy fácil de engañar el espectador del gráfico. Consulte los siguientes dos ejemplos y comentarios sobre este problema (example1, example2 from Junk Charts), así como este artículo de Stephen Few (que concluye: "Ciertamente no puedo concluir, de una vez por todas, que los gráficos con ejes de doble escala nunca son útiles; solo que no puedo pensar en una situación que los justifique a la luz de otras soluciones mejores.") También ver el punto # 4 en esta caricatura ...
Si se determinan, la receta básica es crear su primera gráfica, establecer par(new=TRUE) para evitar que R borre el dispositivo de gráficos, crear la segunda gráfica con axes=FALSE (y establecer xlab y ylab para estar en blanco – ann=FALSE también debería funcionar) y luego usar axis(side=4) para agregar un nuevo eje en el lado derecho, y mtext(...,side=4) para agregar una etiqueta de eje en el lado derecho. Aquí hay un ejemplo usando un poco de datos inventados:
set.seed(101)
x <- 1:10
y <- rnorm(10)
## second data set on a very different scale
z <- runif(10, min=1000, max=10000)
par(mar = c(5, 4, 4, 4) + 0.3) # Leave space for z axis
plot(x, y) # first plot
par(new = TRUE)
plot(x, z, type = "l", axes = FALSE, bty = "n", xlab = "", ylab = "")
axis(side=4, at = pretty(range(z)))
mtext("z", side=4, line=3)
twoord.plot() en el paquete plotrix automatiza este proceso, al igual que doubleYScale() en el latticeExtra paquete.
Otro ejemplo (adaptado de un post de la lista de correo R de Robert W. Baer):
## set up some fake test data
time <- seq(0,72,12)
betagal.abs <- c(0.05,0.18,0.25,0.31,0.32,0.34,0.35)
cell.density <- c(0,1000,2000,3000,4000,5000,6000)
## add extra space to right margin of plot within frame
par(mar=c(5, 4, 4, 6) + 0.1)
## Plot first set of data and draw its axis
plot(time, betagal.abs, pch=16, axes=FALSE, ylim=c(0,1), xlab="", ylab="",
type="b",col="black", main="Mike's test data")
axis(2, ylim=c(0,1),col="black",las=1) ## las=1 makes horizontal labels
mtext("Beta Gal Absorbance",side=2,line=2.5)
box()
## Allow a second plot on the same graph
par(new=TRUE)
## Plot the second plot and put axis scale on right
plot(time, cell.density, pch=15, xlab="", ylab="", ylim=c(0,7000),
axes=FALSE, type="b", col="red")
## a little farther out (line=4) to make room for labels
mtext("Cell Density",side=4,col="red",line=4)
axis(4, ylim=c(0,7000), col="red",col.axis="red",las=1)
## Draw the time axis
axis(1,pretty(range(time),10))
mtext("Time (Hours)",side=1,col="black",line=2.5)
## Add Legend
legend("topleft",legend=c("Beta Gal","Cell Density"),
text.col=c("black","red"),pch=c(16,15),col=c("black","red"))
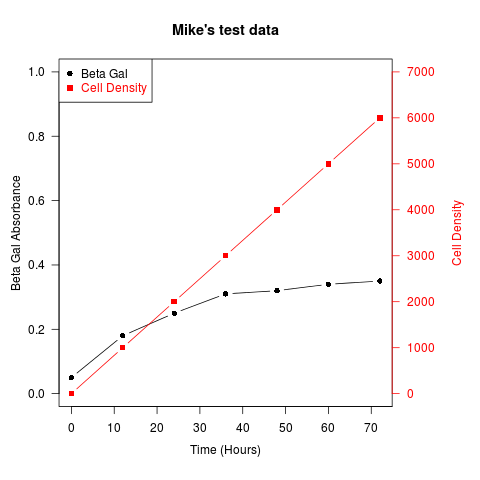
Se pueden usar recetas similares para superponer gráficas de diferentes tipos: gráficas de barras, histogramas, etc..
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/ajaxhispano.com/template/agent.layouts/content.php on line 61
2014-12-16 23:28:05
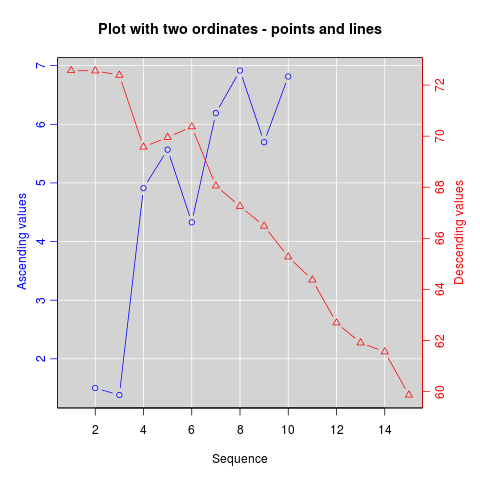
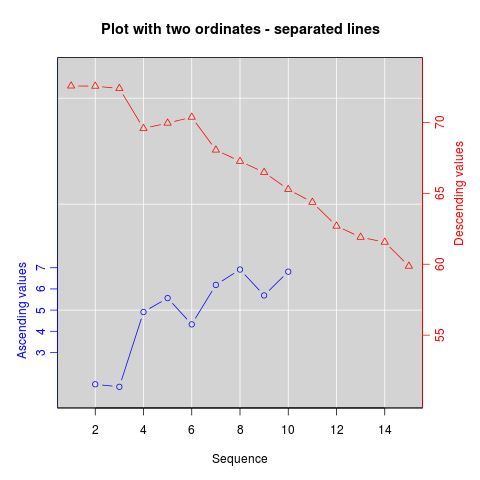
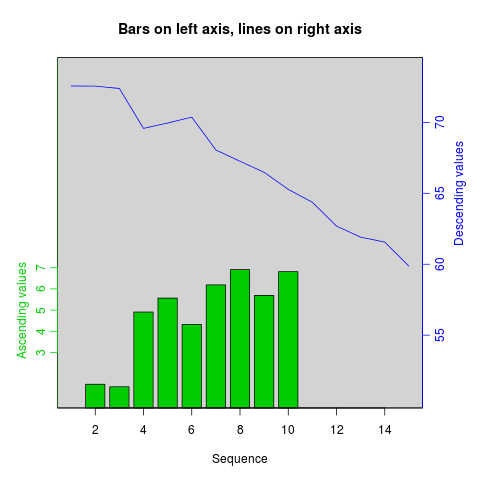
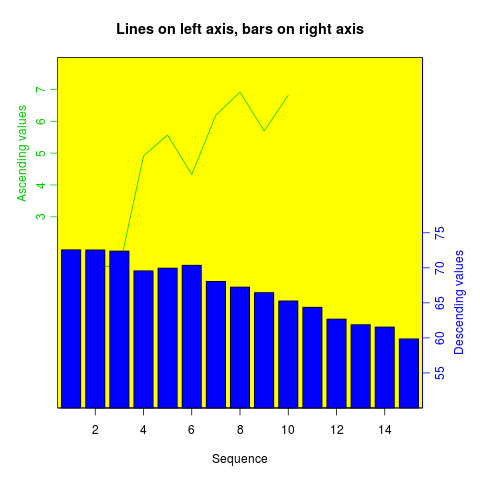
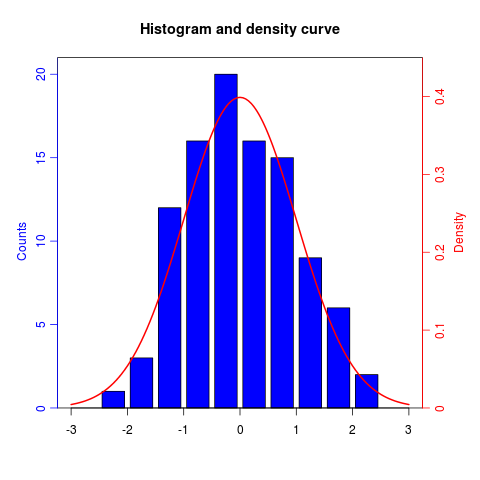
Como su nombre indica, twoord.plot() en el paquete plotrix traza con dos ejes ordenados.
library(plotrix)
example(twoord.plot)
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/ajaxhispano.com/template/agent.layouts/content.php on line 61
2014-09-06 00:30:56
Una opción es hacer dos parcelas una al lado de la otra. ggplot2 proporciona una buena opción para esto con facet_wrap():
dat <- data.frame(x = c(rnorm(100), rnorm(100, 10, 2))
, y = c(rnorm(100), rlnorm(100, 9, 2))
, index = rep(1:2, each = 100)
)
require(ggplot2)
ggplot(dat, aes(x,y)) +
geom_point() +
facet_wrap(~ index, scales = "free_y")
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/ajaxhispano.com/template/agent.layouts/content.php on line 61
2011-05-26 18:00:56
Es una FAQ. Aquí hay una solución más antigua que proporcioné casi hace seis años a la Galería de Gráficos R
Podría mirar, por ejemplo, la función plotVolumeBars() que combina una escala absoluta y una relativa en un gráfico.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/ajaxhispano.com/template/agent.layouts/content.php on line 61
2013-03-20 15:42:19
Si puede renunciar a las etiquetas de escalas/ejes, puede volver a escalar los datos a un intervalo (0, 1). Esto funciona, por ejemplo, para diferentes trakcs 'wiggle' en cromosomas, cuando generalmente estás interesado en correlaciones locales entre las pistas y tienen diferentes escalas (cobertura en miles, Fst 0-1).
# rescale numeric vector into (0, 1) interval
# clip everything outside the range
rescale <- function(vec, lims=range(vec), clip=c(0, 1)) {
# find the coeficients of transforming linear equation
# that maps the lims range to (0, 1)
slope <- (1 - 0) / (lims[2] - lims[1])
intercept <- - slope * lims[1]
xformed <- slope * vec + intercept
# do the clipping
xformed[xformed < 0] <- clip[1]
xformed[xformed > 1] <- clip[2]
xformed
}
Luego, tener un marco de datos con chrom, position, coverage y fst columnas, puedes hacer algo como:
ggplot(d, aes(position)) +
geom_line(aes(y = rescale(fst))) +
geom_line(aes(y = rescale(coverage))) +
facet_wrap(~chrom)
La ventaja de esto es que no estás limitado a dos trakcs.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/ajaxhispano.com/template/agent.layouts/content.php on line 61
2015-04-02 20:15:49
Yo también sugiero, twoord.stackplot() en el paquete plotrix parcelas con más de dos ejes ordenados.
data<-read.table(text=
"e0AL fxAL e0CO fxCO e0BR fxBR anos
51.8 5.9 50.6 6.8 51.0 6.2 1955
54.7 5.9 55.2 6.8 53.5 6.2 1960
57.1 6.0 57.9 6.8 55.9 6.2 1965
59.1 5.6 60.1 6.2 57.9 5.4 1970
61.2 5.1 61.8 5.0 59.8 4.7 1975
63.4 4.5 64.0 4.3 61.8 4.3 1980
65.4 3.9 66.9 3.7 63.5 3.8 1985
67.3 3.4 68.0 3.2 65.5 3.1 1990
69.1 3.0 68.7 3.0 67.5 2.6 1995
70.9 2.8 70.3 2.8 69.5 2.5 2000
72.4 2.5 71.7 2.6 71.1 2.3 2005
73.3 2.3 72.9 2.5 72.1 1.9 2010
74.3 2.2 73.8 2.4 73.2 1.8 2015
75.2 2.0 74.6 2.3 74.2 1.7 2020
76.0 2.0 75.4 2.2 75.2 1.6 2025
76.8 1.9 76.2 2.1 76.1 1.6 2030
77.6 1.9 76.9 2.1 77.1 1.6 2035
78.4 1.9 77.6 2.0 77.9 1.7 2040
79.1 1.8 78.3 1.9 78.7 1.7 2045
79.8 1.8 79.0 1.9 79.5 1.7 2050
80.5 1.8 79.7 1.9 80.3 1.7 2055
81.1 1.8 80.3 1.8 80.9 1.8 2060
81.7 1.8 80.9 1.8 81.6 1.8 2065
82.3 1.8 81.4 1.8 82.2 1.8 2070
82.8 1.8 82.0 1.7 82.8 1.8 2075
83.3 1.8 82.5 1.7 83.4 1.9 2080
83.8 1.8 83.0 1.7 83.9 1.9 2085
84.3 1.9 83.5 1.8 84.4 1.9 2090
84.7 1.9 83.9 1.8 84.9 1.9 2095
85.1 1.9 84.3 1.8 85.4 1.9 2100", header=T)
require(plotrix)
twoord.stackplot(lx=data$anos, rx=data$anos,
ldata=cbind(data$e0AL, data$e0BR, data$e0CO),
rdata=cbind(data$fxAL, data$fxBR, data$fxCO),
lcol=c("black","red", "blue"),
rcol=c("black","red", "blue"),
ltype=c("l","o","b"),
rtype=c("l","o","b"),
lylab="Años de Vida", rylab="Hijos x Mujer",
xlab="Tiempo",
main="Mortalidad/Fecundidad:1950–2100",
border="grey80")
legend("bottomright", c(paste("Proy:",
c("A. Latina", "Brasil", "Colombia"))), cex=1,
col=c("black","red", "blue"), lwd=2, bty="n",
lty=c(1,1,2), pch=c(NA,1,1) )
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/ajaxhispano.com/template/agent.layouts/content.php on line 61
2015-07-29 12:31:48