Cómo trazar dos histogramas juntos en R?
Estoy usando R y tengo dos marcos de datos: zanahorias y pepinos. Cada marco de datos tiene una sola columna numérica que enumera la longitud de todas las zanahorias medidas (total: zanahorias 100k) y pepinos (total: pepinos 50k).
Deseo trazar dos histogramas - longitud de zanahoria y longitud de pepinos - en la misma parcela. Se superponen, así que supongo que también necesito algo de transparencia. También necesito usar frecuencias relativas, no números absolutos, ya que el número de instancias en cada grupo es diferente.
Algo como esto estaría bien, pero no entiendo cómo crearlo a partir de mis dos tablas:
8 answers
Esa imagen a la que se vinculó era para curvas de densidad, no para histogramas.
Si ha estado leyendo en ggplot, entonces tal vez lo único que le falta es combinar sus dos marcos de datos en uno largo.
Así que, vamos a empezar con algo como lo que tienes, dos conjuntos separados de datos y combinarlos.
carrots <- data.frame(length = rnorm(100000, 6, 2))
cukes <- data.frame(length = rnorm(50000, 7, 2.5))
#Now, combine your two dataframes into one. First make a new column in each that will be a variable to identify where they came from later.
carrots$veg <- 'carrot'
cukes$veg <- 'cuke'
#and combine into your new data frame vegLengths
vegLengths <- rbind(carrots, cukes)
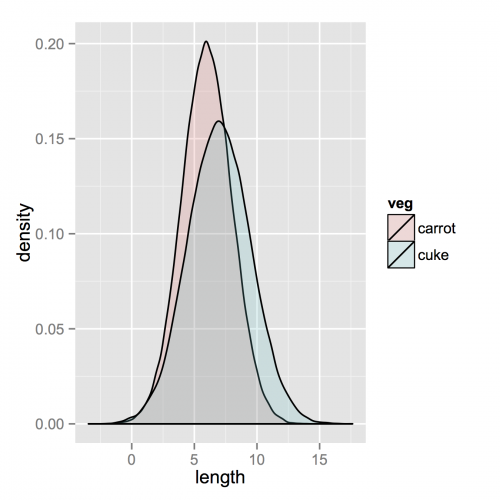
Después de eso, lo cual es innecesario si sus datos ya están en formal largo, solo necesita una línea para hacer su parcela.
ggplot(vegLengths, aes(length, fill = veg)) + geom_density(alpha = 0.2)
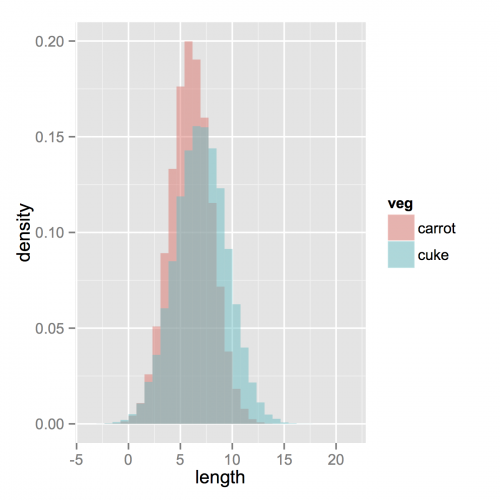
Ahora, si realmente quería histogramas lo siguiente funcionará. Tenga en cuenta que debe cambiar la posición del argumento "pila" predeterminado. Es posible que lo pierda si realmente no tiene una idea de cómo deberían verse sus datos. Un alfa superior se ve mejor allí. También tenga en cuenta que lo hice histogramas de densidad. Es fácil quitar el y = ..density.. para volver a conteos.
ggplot(vegLengths, aes(length, fill = veg)) + geom_histogram(alpha = 0.5, aes(y = ..density..), position = 'identity')
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/ajaxhispano.com/template/agent.layouts/content.php on line 61
2017-06-20 05:37:36
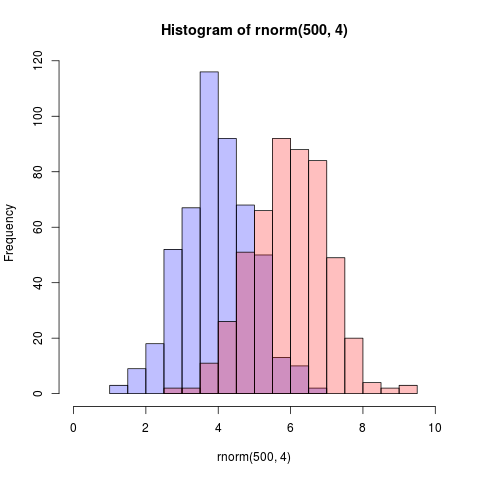
Aquí hay una solución aún más simple que usa gráficos base y mezcla alfa (que no funciona en todos los dispositivos gráficos):
set.seed(42)
p1 <- hist(rnorm(500,4)) # centered at 4
p2 <- hist(rnorm(500,6)) # centered at 6
plot( p1, col=rgb(0,0,1,1/4), xlim=c(0,10)) # first histogram
plot( p2, col=rgb(1,0,0,1/4), xlim=c(0,10), add=T) # second
La clave es que los colores son semitransparentes.
Editar, más de dos años después: Como esto acaba de recibir una votación positiva, me imagino que también podría agregar una imagen visual de lo que produce el código, ya que la mezcla alfa es tan útil:
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/ajaxhispano.com/template/agent.layouts/content.php on line 61
2012-09-21 01:36:46
Aquí hay una función que escribí que utiliza pseudo-transparencia para representar histogramas superpuestos
plotOverlappingHist <- function(a, b, colors=c("white","gray20","gray50"),
breaks=NULL, xlim=NULL, ylim=NULL){
ahist=NULL
bhist=NULL
if(!(is.null(breaks))){
ahist=hist(a,breaks=breaks,plot=F)
bhist=hist(b,breaks=breaks,plot=F)
} else {
ahist=hist(a,plot=F)
bhist=hist(b,plot=F)
dist = ahist$breaks[2]-ahist$breaks[1]
breaks = seq(min(ahist$breaks,bhist$breaks),max(ahist$breaks,bhist$breaks),dist)
ahist=hist(a,breaks=breaks,plot=F)
bhist=hist(b,breaks=breaks,plot=F)
}
if(is.null(xlim)){
xlim = c(min(ahist$breaks,bhist$breaks),max(ahist$breaks,bhist$breaks))
}
if(is.null(ylim)){
ylim = c(0,max(ahist$counts,bhist$counts))
}
overlap = ahist
for(i in 1:length(overlap$counts)){
if(ahist$counts[i] > 0 & bhist$counts[i] > 0){
overlap$counts[i] = min(ahist$counts[i],bhist$counts[i])
} else {
overlap$counts[i] = 0
}
}
plot(ahist, xlim=xlim, ylim=ylim, col=colors[1])
plot(bhist, xlim=xlim, ylim=ylim, col=colors[2], add=T)
plot(overlap, xlim=xlim, ylim=ylim, col=colors[3], add=T)
}
Aquí está otra forma de hacerlo usando el soporte de R para colores transparentes
a=rnorm(1000, 3, 1)
b=rnorm(1000, 6, 1)
hist(a, xlim=c(0,10), col="red")
hist(b, add=T, col=rgb(0, 1, 0, 0.5) )
Los resultados terminan pareciendo algo como esto:
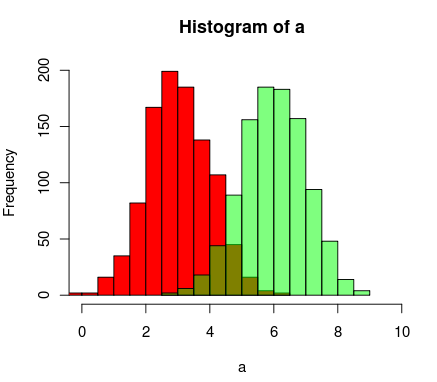
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/ajaxhispano.com/template/agent.layouts/content.php on line 61
2013-07-05 01:59:16
Ya hay respuestas hermosas, pero pensé en agregar esto. A mí me parece bien.
(Copiado números aleatorios de @ Dirk). library(scales) es necesario`
set.seed(42)
hist(rnorm(500,4),xlim=c(0,10),col='skyblue',border=F)
hist(rnorm(500,6),add=T,col=scales::alpha('red',.5),border=F)
El resultado es...
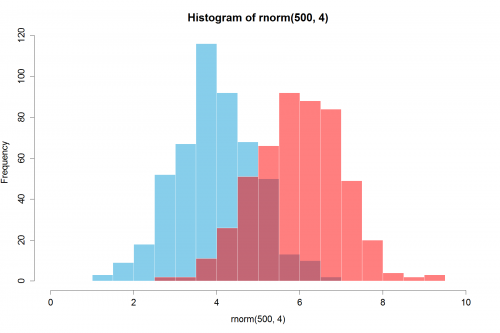
Actualización: Este superposición función también puede ser útil para algunos.
hist0 <- function(...,col='skyblue',border=T) hist(...,col=col,border=border)
Siento que el resultado de {[5] } es más bonito de mirar que hist
hist2 <- function(var1, var2,name1='',name2='',
breaks = min(max(length(var1), length(var2)),20),
main0 = "", alpha0 = 0.5,grey=0,border=F,...) {
library(scales)
colh <- c(rgb(0, 1, 0, alpha0), rgb(1, 0, 0, alpha0))
if(grey) colh <- c(alpha(grey(0.1,alpha0)), alpha(grey(0.9,alpha0)))
max0 = max(var1, var2)
min0 = min(var1, var2)
den1_max <- hist(var1, breaks = breaks, plot = F)$density %>% max
den2_max <- hist(var2, breaks = breaks, plot = F)$density %>% max
den_max <- max(den2_max, den1_max)*1.2
var1 %>% hist0(xlim = c(min0 , max0) , breaks = breaks,
freq = F, col = colh[1], ylim = c(0, den_max), main = main0,border=border,...)
var2 %>% hist0(xlim = c(min0 , max0), breaks = breaks,
freq = F, col = colh[2], ylim = c(0, den_max), add = T,border=border,...)
legend(min0,den_max, legend = c(
ifelse(nchar(name1)==0,substitute(var1) %>% deparse,name1),
ifelse(nchar(name2)==0,substitute(var2) %>% deparse,name2),
"Overlap"), fill = c('white','white', colh[1]), bty = "n", cex=1,ncol=3)
legend(min0,den_max, legend = c(
ifelse(nchar(name1)==0,substitute(var1) %>% deparse,name1),
ifelse(nchar(name2)==0,substitute(var2) %>% deparse,name2),
"Overlap"), fill = c(colh, colh[2]), bty = "n", cex=1,ncol=3) }
El resultado de
par(mar=c(3, 4, 3, 2) + 0.1)
set.seed(100)
hist2(rnorm(10000,2),rnorm(10000,3),breaks = 50)
Es
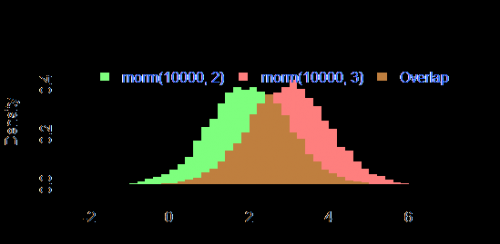
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/ajaxhispano.com/template/agent.layouts/content.php on line 61
2015-12-03 19:20:08
Aquí hay un ejemplo de cómo puede hacerlo en gráficos R "clásicos":
## generate some random data
carrotLengths <- rnorm(1000,15,5)
cucumberLengths <- rnorm(200,20,7)
## calculate the histograms - don't plot yet
histCarrot <- hist(carrotLengths,plot = FALSE)
histCucumber <- hist(cucumberLengths,plot = FALSE)
## calculate the range of the graph
xlim <- range(histCucumber$breaks,histCarrot$breaks)
ylim <- range(0,histCucumber$density,
histCarrot$density)
## plot the first graph
plot(histCarrot,xlim = xlim, ylim = ylim,
col = rgb(1,0,0,0.4),xlab = 'Lengths',
freq = FALSE, ## relative, not absolute frequency
main = 'Distribution of carrots and cucumbers')
## plot the second graph on top of this
opar <- par(new = FALSE)
plot(histCucumber,xlim = xlim, ylim = ylim,
xaxt = 'n', yaxt = 'n', ## don't add axes
col = rgb(0,0,1,0.4), add = TRUE,
freq = FALSE) ## relative, not absolute frequency
## add a legend in the corner
legend('topleft',c('Carrots','Cucumbers'),
fill = rgb(1:0,0,0:1,0.4), bty = 'n',
border = NA)
par(opar)
El único problema con esto es que se ve mucho mejor si los saltos del histograma están alineados, lo que puede tener que hacerse manualmente (en los argumentos pasados a hist).
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/ajaxhispano.com/template/agent.layouts/content.php on line 61
2015-01-28 02:42:58
Aquí está la versión como la de ggplot2 que di solo en base R. Copié algunas de @nullglob.
Generar los datos
carrots <- rnorm(100000,5,2)
cukes <- rnorm(50000,7,2.5)
No es necesario ponerlo en un marco de datos como con ggplot2. El inconveniente de este método es que usted tiene que escribir mucho más de los detalles de la trama. La ventaja es que usted tiene el control sobre más detalles de la trama.
## calculate the density - don't plot yet
densCarrot <- density(carrots)
densCuke <- density(cukes)
## calculate the range of the graph
xlim <- range(densCuke$x,densCarrot$x)
ylim <- range(0,densCuke$y, densCarrot$y)
#pick the colours
carrotCol <- rgb(1,0,0,0.2)
cukeCol <- rgb(0,0,1,0.2)
## plot the carrots and set up most of the plot parameters
plot(densCarrot, xlim = xlim, ylim = ylim, xlab = 'Lengths',
main = 'Distribution of carrots and cucumbers',
panel.first = grid())
#put our density plots in
polygon(densCarrot, density = -1, col = carrotCol)
polygon(densCuke, density = -1, col = cukeCol)
## add a legend in the corner
legend('topleft',c('Carrots','Cucumbers'),
fill = c(carrotCol, cukeCol), bty = 'n',
border = NA)
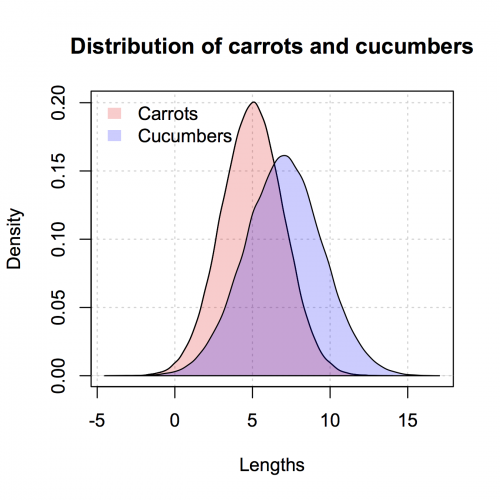
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/ajaxhispano.com/template/agent.layouts/content.php on line 61
2013-10-15 01:56:27
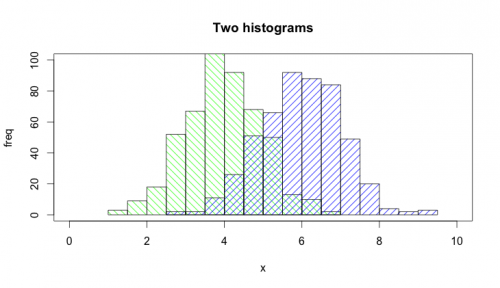
@Dirk Eddelbuettel: La idea básica es excelente, pero el código como se muestra se puede mejorar. [Toma mucho tiempo explicar, por lo tanto una respuesta separada y no un comentario.]
La función hist() por defecto dibuja gráficos, por lo que debe agregar la opción plot=FALSE. Además, es más claro establecer el área de la gráfica mediante una llamada plot(0,0,type="n",...) en la que puede agregar las etiquetas del eje, el título de la gráfica, etc. Por último, me gustaría mencionar que también se podría utilizar el sombreado para distinguir entre los dos histogramas. Aquí está el código:
set.seed(42)
p1 <- hist(rnorm(500,4),plot=FALSE)
p2 <- hist(rnorm(500,6),plot=FALSE)
plot(0,0,type="n",xlim=c(0,10),ylim=c(0,100),xlab="x",ylab="freq",main="Two histograms")
plot(p1,col="green",density=10,angle=135,add=TRUE)
plot(p2,col="blue",density=10,angle=45,add=TRUE)
Y aquí está el resultado (un poco demasiado amplio debido a RStudio: -)):
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/ajaxhispano.com/template/agent.layouts/content.php on line 61
2014-10-09 15:18:43
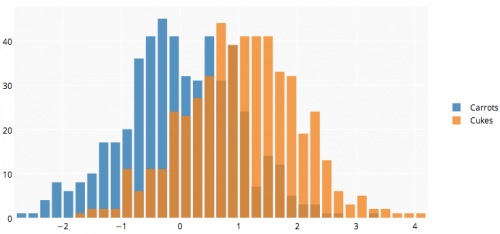
La API R de Plotly podría ser útil para usted. El siguiente gráfico es aquí.
library(plotly)
#add username and key
p <- plotly(username="Username", key="API_KEY")
#generate data
x0 = rnorm(500)
x1 = rnorm(500)+1
#arrange your graph
data0 = list(x=x0,
name = "Carrots",
type='histogramx',
opacity = 0.8)
data1 = list(x=x1,
name = "Cukes",
type='histogramx',
opacity = 0.8)
#specify type as 'overlay'
layout <- list(barmode='overlay',
plot_bgcolor = 'rgba(249,249,251,.85)')
#format response, and use 'browseURL' to open graph tab in your browser.
response = p$plotly(data0, data1, kwargs=list(layout=layout))
url = response$url
filename = response$filename
browseURL(response$url)
Revelación completa: Estoy en el equipo.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/ajaxhispano.com/template/agent.layouts/content.php on line 61
2014-03-26 05:25:29